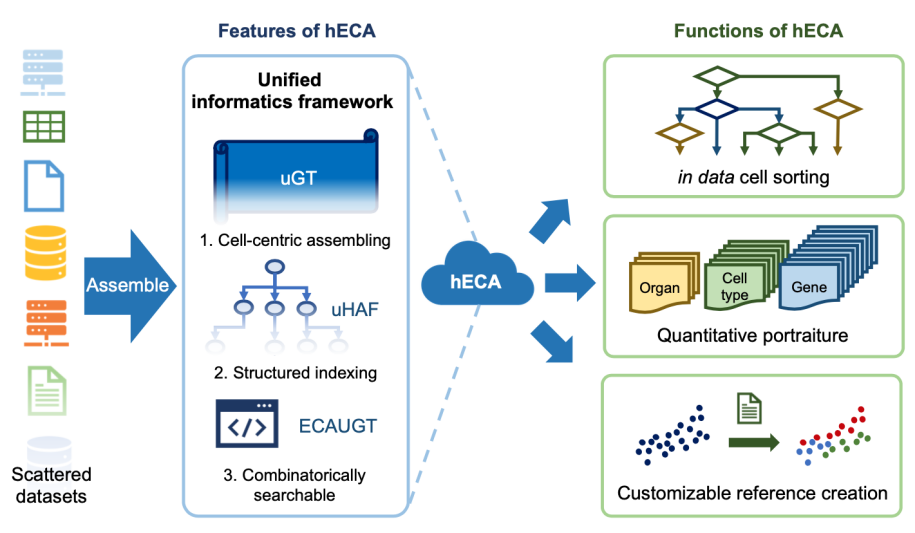
1. 首个细胞级组装的人体集成细胞图谱hECA(张学工团队)
2021年9月和2022年5月,清华大学张学工团队先后在《国家科学评论》(National Science Review)发表观点文章“迈向细胞图谱组装的统一信息框架”和在Cell旗下iScience发表题为hECA: the cell-centric assembly of a cell atlas的文章,提出了一种能够无缝组装海量单细胞数据的统一信息学框架,并基于此框架建成了首个细胞级组装的人类集成细胞图谱human Ensemble Cell Atlas(hECA)。hECA涵盖了来自116个公开数据集、包括38个器官和11个系统的109万个人体细胞,这些细胞的基因表达等信息被统一存储到了一个支持超宽超深数据存储、管理和索引的数据库系统之中,形成统一的细胞信息表,可实现秒级的数据快速检索查询,突破了细胞图谱研究中数据分散、整合困难等关键问题。在该框架下,文章进一步提出了“数基细胞筛选”、“多生物学实体全息画像”等细胞图谱应用新范式,为未来细胞图谱建设和单细胞技术的研究与应用提供了全新的研究思路和方法工具。
科普网页链接 《国家科学评论》公众号: https://mp.weixin.qq.com/s/Zt_hhPZP8WL4QsgjdbYa8Q 《细胞科学》公众号: https://mp.weixin.qq.com/s/Sl8tpD0UlewmuL2ZRSyHQg 《测序中国》公众号: https://mp.weixin.qq.com/s/eCY605JMrc1pLdc32heNAg
2. 包虫病AI诊断新技术(张学工团队)
2023年9月26日,清华大学自动化系、北京信息科学与技术国家研究中心张学工教授团队与神经调控国家工程研究中心团队、青海大学附属医院樊海宁教授团队等合作,在《柳叶刀-数字健康》(The Lancet Digital Health)杂志发表了最新研究成果。他们开发的名为EDAM的包虫病辅助诊断AI系统,用普通平扫CT图像实现包虫病的自动检测和分型,为因医生资源短缺而包虫病高发的西部地区提供了筛查包虫病的AI诊断新技术,有望彻底解决长期困扰高原牧区的包虫病筛查难题。

科普网页链接 《清华大学自动化系》公众号: https://mp.weixin.qq.com/s/KzdVsZnfDwB-2H3byXa8Cw 《柳叶刀TheLancet》公众号: https://mp.weixin.qq.com/s/1uYK6W9tZjuGRIkeBcFjkg
3. 基于CT影像的肺癌瘤内异质性量化与可视化新方法(张学工团队)
2022年7月24日,清华大学自动化系、北京信息科学与技术国家研究中心张学工教授团队与广东省人民医院钟文昭教授团队合作,在放射影像学国际知名期刊《欧洲放射学》(European Radiology)发表了最新研究成果。他们创新性地提出了一种基于CT影像的肿瘤内异质性可视化和量化的方法—ITHscore,并发现ITHscore与肿瘤演进进程存在相关性,可用于肿瘤预后预测,有望用于临床指导术式选择或药物干预时机与方案抉择。

科普网页链接 《肺癌前沿》公众号: https://mp.weixin.qq.com/s/7Y_b6u6-sMQNhhoJMJlbmw
4. 揭示半月板不同分区退变微环境中的细胞特征(张学工团队)
2022年12月22日,清华大学自动化系、北京信息科学与技术国家研究中心张学工教授团队与四川大学华西医院骨科运动医学中心付维力副教授团队合作在国际著名生物学综合期刊eLife发表了最新研究成果。他们利用单细胞测序结合多重免疫荧光的方法,构建了健康状态以及退变状态下半月板红、白区的细胞图谱,为研究半月板内微环境稳态以及探索骨关节炎的发生机制提供重要依据。

科普网页链接 《蛋白质组代谢组学》公众号: https://mp.weixin.qq.com/s/JxEAdsQUQT8LKPLNDOOabg
5. 首次揭示白血病微小残留病的本质(古槿团队)
2022年2月10日,中国医学科学院血液病医院(中国医学科学院血液学研究所)竺晓凡、程涛团队和清华大学古槿团队在国际著名期刊Nature Cell Biology上发表最新研究成果。他们以儿童B细胞急性淋巴细胞白血病这一儿童最常见的血液系统恶性肿瘤为研究对象,在国际上首次利用单细胞测序技术绘制了儿童B-ALL MRD的单细胞图谱,发现低氧信号通路异常激活是儿童B-ALL MRD细胞的重要特征,低氧信号通路是儿童复发难治B-ALL治疗的潜在治疗靶点。
科普网页链接 《BioArt》公众号: https://baijiahao.baidu.com/s?id=1724427552460973251&wfr=spider&for=pc
6. 揭示尿素循环关键酶CPS1缺失引起代谢重编程诱导肝细胞癌发生的新机制(古槿团队)
2021年8月3日,海军军医大学王红阳院士、陈磊研究员与清华大学古槿教授团队在Hepatology杂志发表了最新研究成果。该论文通过大数据挖掘定义了CPS1缺失肝癌亚型,利用质谱检测揭示肝癌细胞内脂肪酸氧化(FAO)代谢活跃,进而通过活化转录因子FOXM1促进肿瘤发生,并借助类器官提出针对此类肝癌亚型的靶向治疗新策略。

科普网页链接 《BioArt》公众号: https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652547744&idx=4&sn=b92ed62a720dc6af339a9dace481177c&chksm=84e13914b396b002c089f5c2c7d8b887bf1e3fefdec2e98303c6224109c8f2f8bd11664fc6b3&scene=27
7. 绘制原发性肝癌高分辨率空间转录组图谱(古槿团队)
2021年12月17日,海军军医大学国家肝癌科学中心王红阳院士、陈磊研究员团队联合清华大学古槿教授团队在国际著名期刊Science Advances发表研究成果。他们成功绘制了原发性肝癌高分辨率空间转录组图谱,并确定了肿瘤微环境特征。同时研究团队提出了一种TLS-50标记,用于在空间上精确定位三级淋巴样结构(tls),并揭示了tls的独特组成是由它们与肿瘤细胞的距离决定的。上述研究发现为肝癌复杂的生态系统提供了新的见解,并有潜力改善个体化的癌症预防和药物发现。
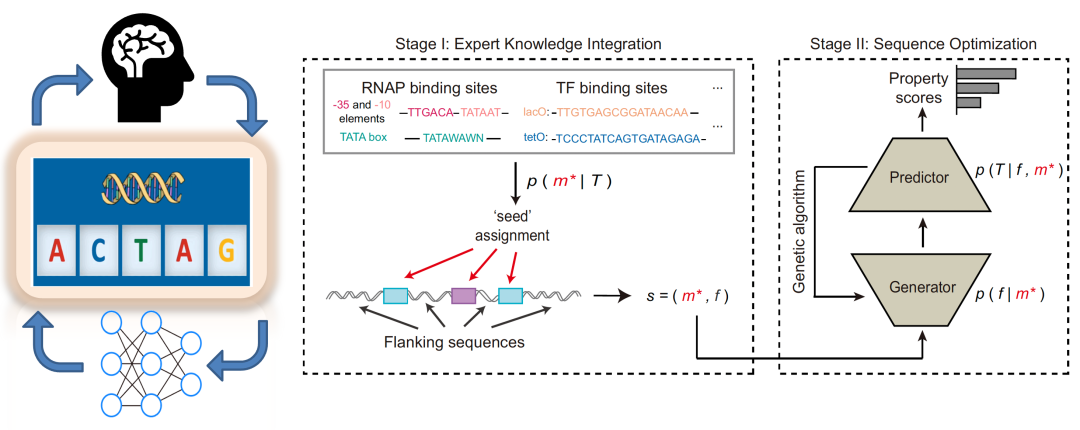
8. 知识引导与数据驱动相融合的合成启动子AI设计方法(汪小我团队)
2023年10月9日,清华大学自动化系汪小我团队在《自然-通讯》(Nature Communications)期刊上发表了最新研究成果。他们提出了一种将专家知识与大数据学习相融合的合成启动子人工智能辅助设计方法,发现了转录因子结合位点旁侧序列在启动子优化设计中的重要作用,为突破基因调控元件设计中面临的高维度、小样本的核心难题提供了新的思路。
科普网页链接 《测序中国》公众号: https://mp.weixin.qq.com/s/T8f59oh8ff0CDVESZBAc0Q 《BioArt》公众号: https://mp.weixin.qq.com/s/EcfmqlihLE9JHsDstBqW2A 《清华大学自动化系》公众号: https://mp.weixin.qq.com/s/KNVrVTzDIWClKvl5aIKY7A
9. 预测早期肿瘤的人工智能算法DISMIR(汪小我团队)
2021年7月9日,清华大学自动化系汪小我教授团队在知名期刊Briefings in Bioinformatics上发表了最新研究成果。在生物体中,伴随着细胞凋亡等过程,细胞内的DNA片段会被释放到血浆中成为游离DNA (cfDNA)。在癌症发生早期,当患者还未表现出明显的临床症状时,细胞内DNA的状态就已经发生了变化,这些DNA被释放到血浆中,使得血浆cfDNA中蕴含了与癌症相关的重要信息。基于深度学习方法,该研究实现了通过血浆cfDNA的WGBS数据进行高精度的癌症检测,且在超低测序深度下仍旧保持较好的检测效果。

科普网页链接 《甲基化》公众号: https://mp.weixin.qq.com/s/uo_4gPhw0UcAc2Se8QUYAw
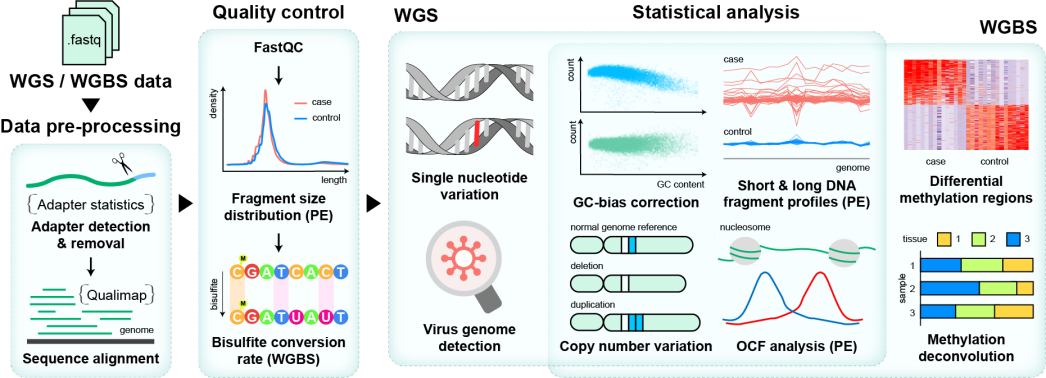
10. 细胞游离DNA测序数据一体化分析流程软件包cfDNApip(汪小我团队)
2021年5月27日,清华大学自动化系汪小我教授团队和清华–福州数据技术研究院团队合作,在Bioinformatics上发表了最新研究。他们开发了一套系统的针对cfDNA全基因组测序(WGS)和全基因组甲基化测序(WGBS)的数据分析软件包“cfDNApipe”。cfDNApipe面向实际的科研和临床应用需求,以简单易用的自动化数据分析为设计目标,帮助研究人员克服数据分析流程中的困难,可得到系统、准确的cfDNA测序数据分析结果。
科普网页链接 《中国抗癌协会肿瘤标志专业委员会》公众号: https://mp.weixin.qq.com/s/rjFQ74gXe3Nc3KnlCIL-UA
11. 自动归纳基因调控序列编码模式的神经网络解释方法(汪小我团队)
2023年4月6日,清华大学自动化系汪小我团队与美国斯坦福大学统计系王永雄团队在《美国科学院院刊》(PNAS)发表最新研究成果。他们提出自动归纳基因调控序列编码模式的神经网络解释方法。该方法针对广泛应用于基因调控序列研究的卷积神经网络(CNN)模型,通过剖析神经网络中的多面神经元机制(multifaceted neuron),开发了神经网络解释算法(NeuronMotif),实现了从神经元中自动归纳和提取转录因子结合位点等关键序列模式(Motif)及其组合、次序、间距等基因调控序列编码规则。该方法能够帮助研究者更加深入地理解基因调控编码规律,同时为神经网络模型的解释提供了新手段。

科普网页链接 《测序中国》公众号: https://mp.weixin.qq.com/s/NC_xsG6XmKBPKDmfIK1fkg 《清华大学自动化系》公众号: https://mp.weixin.qq.com/s/zd1BOY1m4k5OeOE4yFdvxg
12. 揭示细胞衰老过程的基因组三维结构改变规律(汪小我团队)
2021年6月17日,清华大学汪小我教授与颉伟教授合作在Genome Research发表最新研究成果。该工作通过多组学大数据的生物信息整合分析,发现了细胞衰老过程中异染色质的大规模丢失伴随着染色质开放性和多层次的染色质三维结构的变化,并且在异染色质丢失区域出现了异常基因表达泄露(gene expression leakage)的现象。这一研究丰富了我们对衰老细胞异染色质状态的认识,建立了衰老细胞中异染色质开放性、三维基因组结构、转录调控之间的联系。这些发现对于后续识别衰老细胞和干预衰老过程的研究有着重要参考意义。

科普网页链接 《BioArt》公众号: https://mp.weixin.qq.com/s/z73388oopokPxQLSteslqg
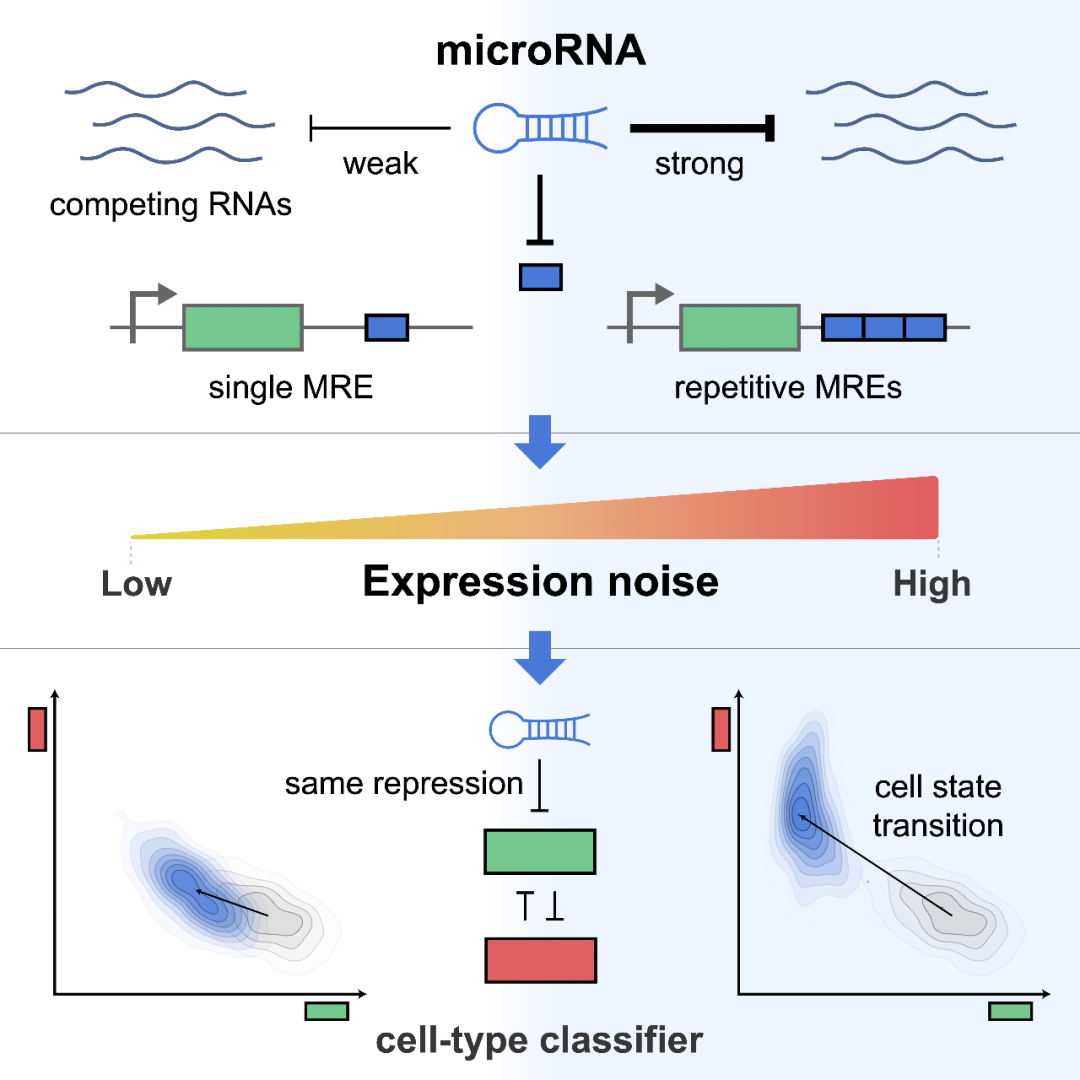
13. 揭示microRNA降噪机制,指导构建基因回路实现肿瘤细胞精准识别(汪小我团队)
2021年8月24日,清华大学自动化系汪小我教授团队在国际著名期刊Cell Reports上发表了最新研究成果。该研究基于“数基–碳基融合”的思路,利用数学建模仿真和合成生物学实验观测,揭示了microRNA的基因表达降噪机制,并将其应用于合成基因回路设计,构建了人工基因细胞分类器,实现了对肿瘤细胞的精准识别。
科普网页链接 《小柯生命》公众号: https://mp.weixin.qq.com/s/pLSdT70AJ0B8UPgw5p6Qzg 《清华大学自动化系》公众号: https://mp.weixin.qq.com/s/KlX5yhX8TGrJjMDV0V_RUg
14. 揭示血液游离核糖体DNA甲基化谱可用于癌症诊断(汪小我团队)
2022年7月9日,清华大学汪小我教授团队和解放军总医院第五医学中心陆荫英教授团队联合攻关,在Briefings in Bioinformatics杂志发表重要成果。研究团队设计了一种新的比对策略用于研究rDNA甲基化,并分析了各种生理病理样本中单CpG分辨率下的甲基化模式。该研究拓宽了人们对rDNA调控的理解,并提出了血液游离rDNA甲基化谱作为疾病生物标志物的潜在用途。
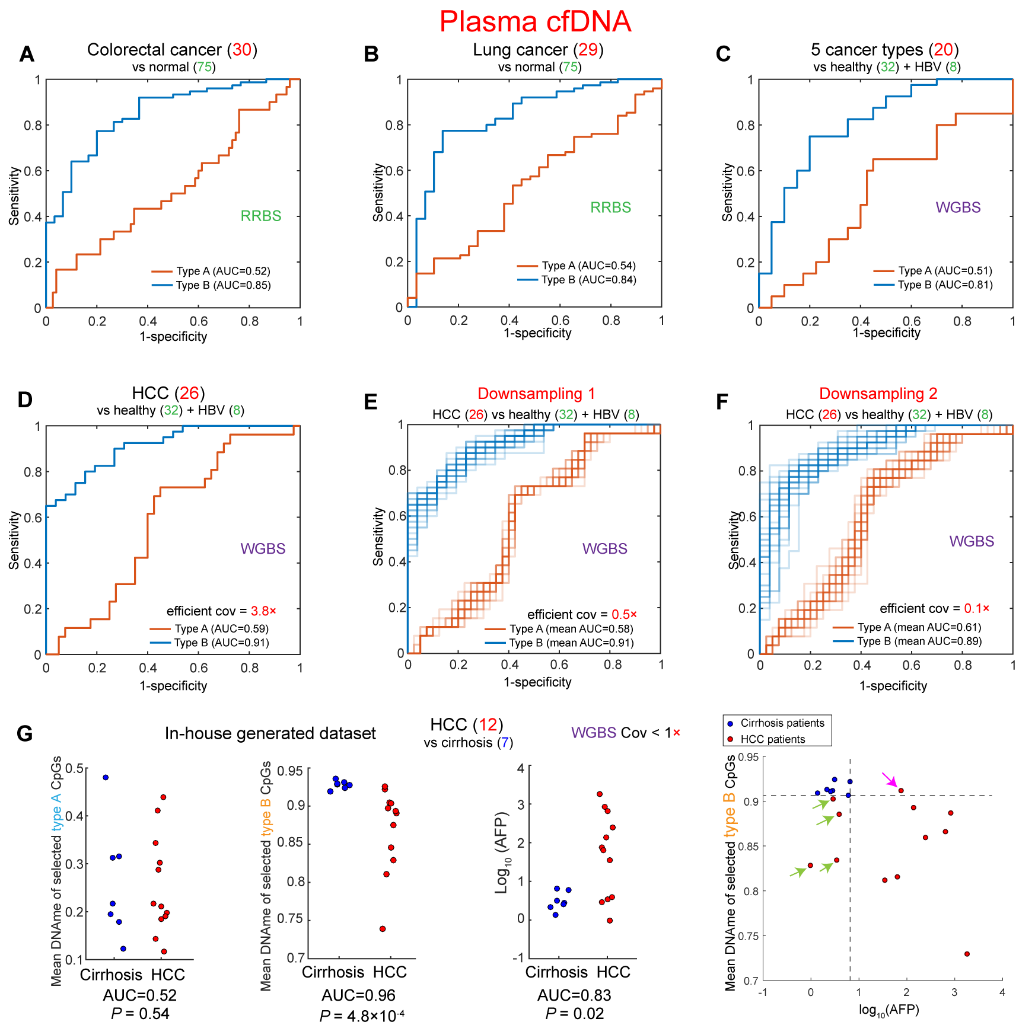
科普网页链接 《测序中国》公众号: https://mp.weixin.qq.com/s/0UudcNhYKbnA6cpS7ETxsQ
15. 首次用人工智能方法设计产生全新的基因启动子(汪小我团队)
2020年5月19日,清华大学汪小我课题组题在Nucleic acids research杂志上以“突破性研究论文(Breakthrough Article)”形式发表最新研究成果。该研究将人工智能技术应用于构建全新的基因调控元件,从自动化设计的角度,利用深度学习技术并融合生物先验知识来建立调控元件的生成模型。通过在计算机中的算法寻优部分替代生物实验上的随机搜索,可以大大提高实验的成功率。该项研究首次用人工智能方法设计产生全新的基因启动子,为生物调控元件的设计和优化提供了崭新的手段。
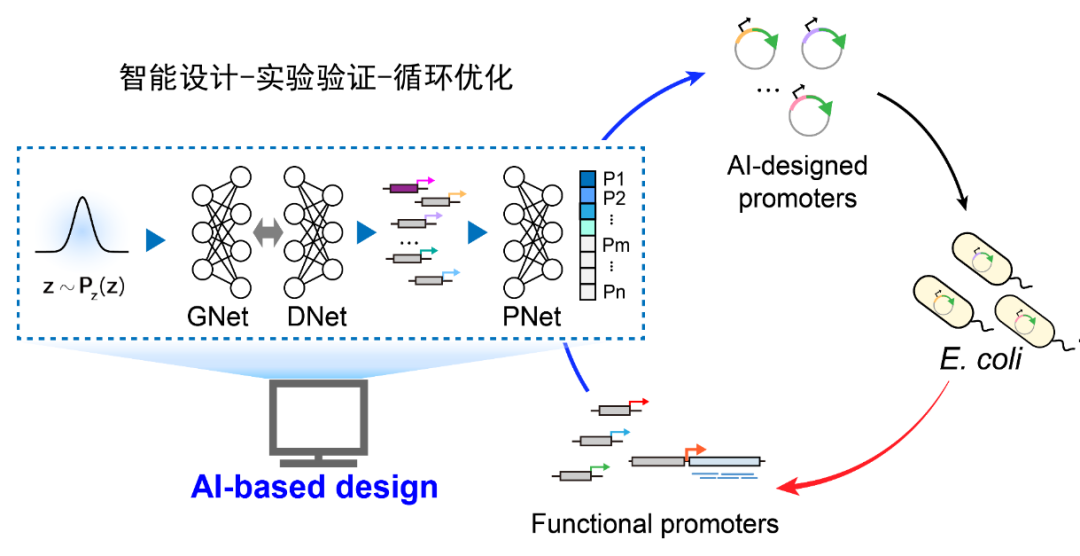
科普网页链接 《BioArt》公众号: https://mp.weixin.qq.com/s/l-ttGpNg9JYRQo6eLVsDFw
16. 实现环状RNA的可编程翻译调控,精准识别目标细胞类型(谢震团队)
2023年11月8日,清华大学自动化系、北京信息科学与技术国家研究中心谢震课题组在《自然-通讯》(Nature Communications)期刊上发表了最新研究成果。他们开发了一种核糖体内部进入位点(internal ribosome entry site, IRES)的工程改造技术(PROMITAR),实现了仅用单个环状 RNA 分子即可感应多种细胞内源微小 RNA(miRNA)信号,控制 IRES 元件在特定细胞中的翻译活性。
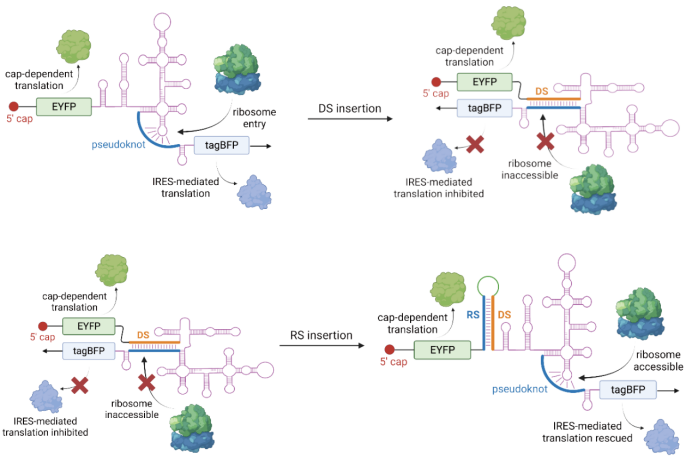
科普网页链接 《合成生物学期刊》公众号: https://mp.weixin.qq.com/s/XPqkF0mZ1ke_Dq0Pqy4N6Q 《生辉SynBio》公众号: https://mp.weixin.qq.com/s/1xw1Dg6dXfe_vN4M2wiunw
17. 从DNA代谢通路角度探究引导编辑的影响因素(谢震团队)
2023年7月3号,清华大学自动化系谢震团队在Synthetic and Systems Biotechnology期刊上发表了最新研究成果。研究探究了非MMR途径的FEN1、LIG1以及MMR途径中的EXO1、MLH1四个基因在HEK293FT细胞系中对PE编辑效率的影响,并验证了MLH1基因是限制PE编辑过程的关键因素。
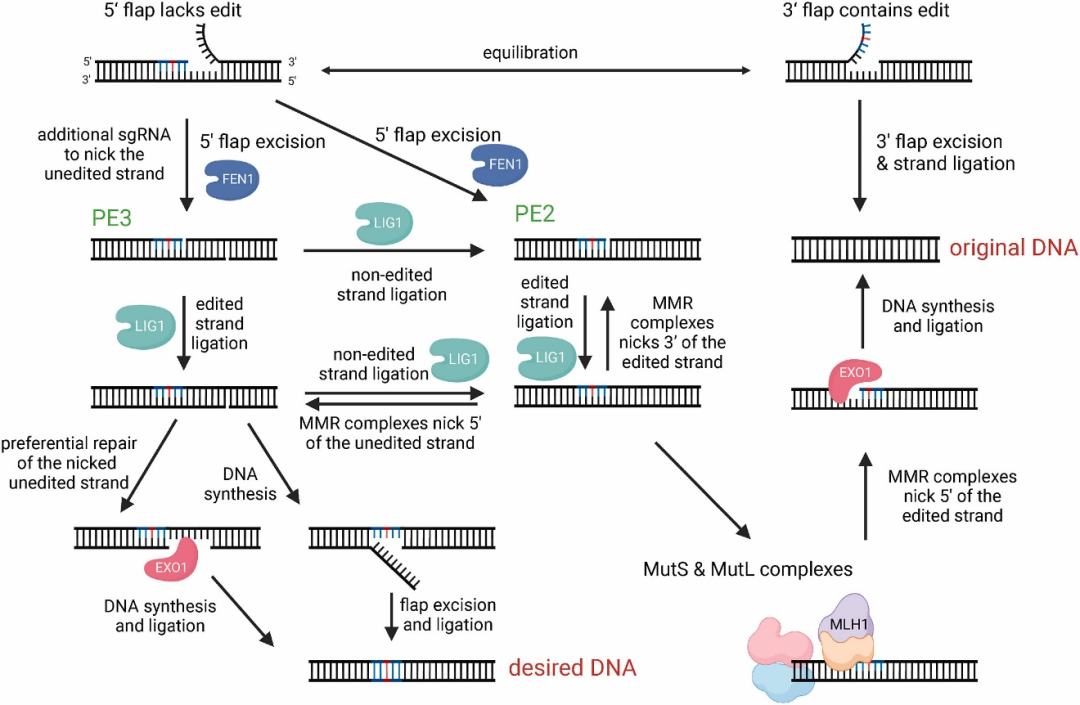
科普网页链接 《合成和系统生物技术》公众号: https://mp.weixin.qq.com/s/vXQnDKORqVbpe7JTYPWczg
18. 中晚期下咽癌临床分层诊疗预测模型(谢震团队)
2023年3月16日,清华大学自动化系、北京信息科学与技术国家研究中心谢震课题组与北京同仁医院黄志刚、张洋团队合作在在《自然-通讯》(Nature Communications)期刊上发表了最新研究成果。他们基于中晚期下咽癌的临床样本,利用单细胞转录组测序和混池转录组测序技术,通过系统的生物信息学分析,绘制了首张高分辨率的下咽癌肿瘤微环境细胞图谱,筛选出了与预后密切相关的肿瘤细胞共享功能模块(基因集),并建立了基于微环境非恶性肿瘤细胞亚型构成的治疗响应预测模型,为中晚期下咽癌的临床分层诊疗提供了新方案。

科普网页链接 《BioArt》公众号: https://mp.weixin.qq.com/s/phnZrRGS2pXLWA6MO8arLw
19. 大幅提高小Cas9 PAM 识别范围的新技术(谢震团队)
2019年2月4日,清华大学谢震课题组在《自然·通讯》(Nature Communications)发表最新研究成果。他们利用进化信息,采用嵌合Cas9的思路,将 SaCas9的 PAM 范围进一步大幅度提高,最终靶向超过1/4的序列。

科普网页链接 《BioArt》公众号: https://mp.weixin.qq.com/s/mHrASzJkAUhhAaFOo4pFXA
20. 实现“编程改造”溶瘤病毒,“专杀”肿瘤细胞(谢震团队)
2019 年 10 月 22 日,清华大学北京信息科学与技术国家研究中心谢震课题组在《自然·通讯》(Nature Communications)发表最新研究。他们构建了模块化的合成基因线路,调控溶瘤腺病毒在肿瘤细胞中选择性复制,从而特异性杀伤肿瘤细胞、刺激抗肿瘤免疫。该研究为溶瘤腺病毒的精准工程化改造提供了新型解决方案,提高了溶瘤病毒靶向肿瘤免疫治疗的效果和安全性。
科普网页链接 《 DeepTech》公众号: https://mp.weixin.qq.com/s/CCjDqV-lNCpixq3UUowbqg
21. 单细胞表观基因组学分析工具数据库scEpiTools(江瑞团队)
2023年9月27日,清华大学自动化系江瑞教授团队在Journal of Genetics and Genomics期刊发表了最新研究成果。该研究建立了一个单细胞表观基因组学分析工具数据库scEpiTools,收集整理并详细注释了622篇相关的方法类、综述类、测序技术及应用类论文,并将其归纳为14种主类别和93种子类别,提供包括检索、推荐、分层次浏览、在线分析在内的多种功能,有望成为单细胞表观基因组学研究的重要资源和工具。
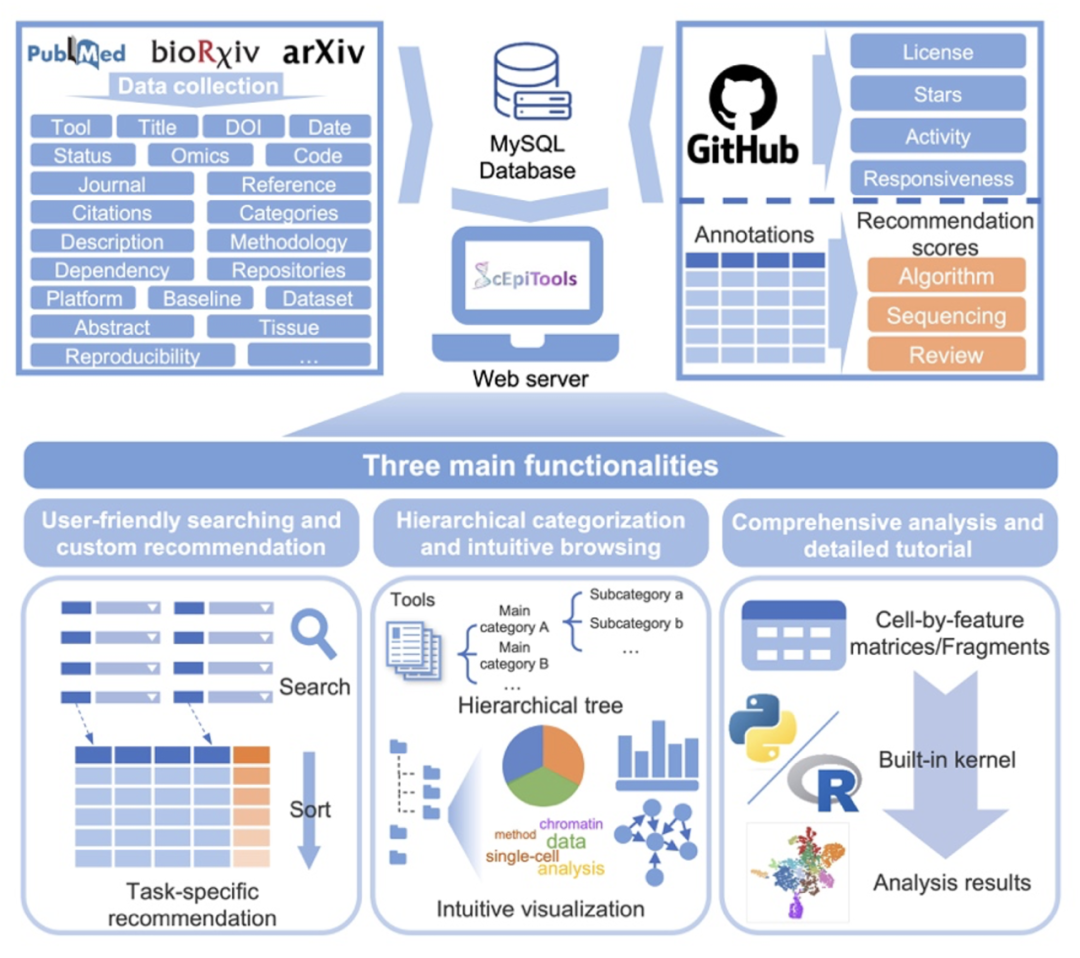
科普网页链接 《JGG 遗传学报》公众号: https://mp.weixin.qq.com/s/FtSV2NSku5QYcqefomPdJg
22. 首个针对单细胞染色质开放性数据的细胞类型辨识神经网络模型EpiAnno(江瑞团队)
2022年2月10日,清华大学自动化系江瑞教授团队在国际顶级期刊Nature Machine Intelligence发表了最新研究成果。提出了整合细胞图谱与单细胞染色质开放性数据(scCAS)辨识细胞类型的贝叶斯神经网络模型EpiAnno。该模型不仅能以极高精度辨识数据中的细胞类型,还能有效提取细胞类型的特征,应用于基因和信号通路富集分析、致病遗传因素识别等生物医学研究中。
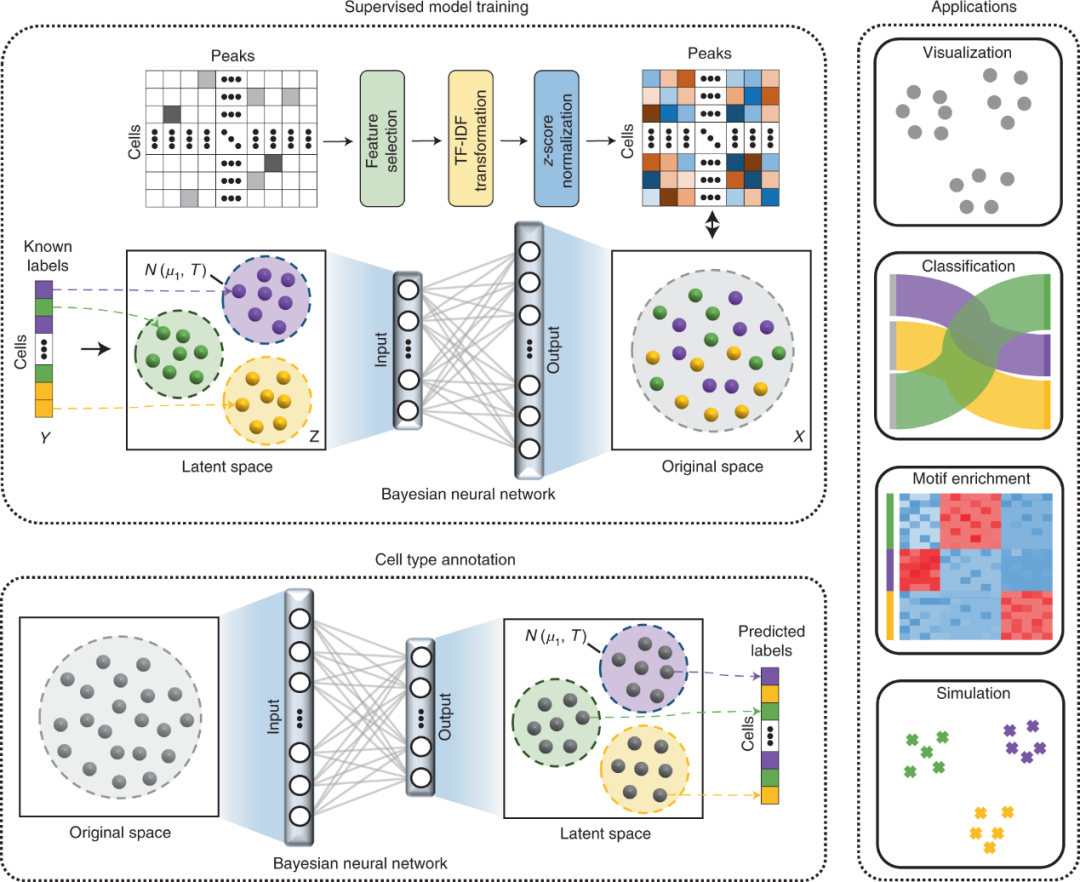
科普网页链接 《测序中国》公众号: https://mp.weixin.qq.com/s/D3ZsXcIyOxHsyaXUWf56Hg
23. 全基因功能注释HiChIP数据库(江瑞团队)
2022年10月10日,美国科学院院士、斯坦福大学统计系Wing Hung Wong教授课题组联合清华大学自动化系江瑞课题组在Nucleic Acids Research上发表最新成果。研究发表了首个附带全基因组功能注释HiChIP数据库。数据库收录了截止于2022年5月份上传至GEO数据库的人类HiChIP约200个样本。HiChIP数据库采用了统一的数据处理流程,从原始Fastq测序原始数据统一处理至不同分辨率的HiChIP相互作用数据。HiChIP数据库覆盖了超过100个人类细胞类型下总计2.62亿条HiChIP相互作用。
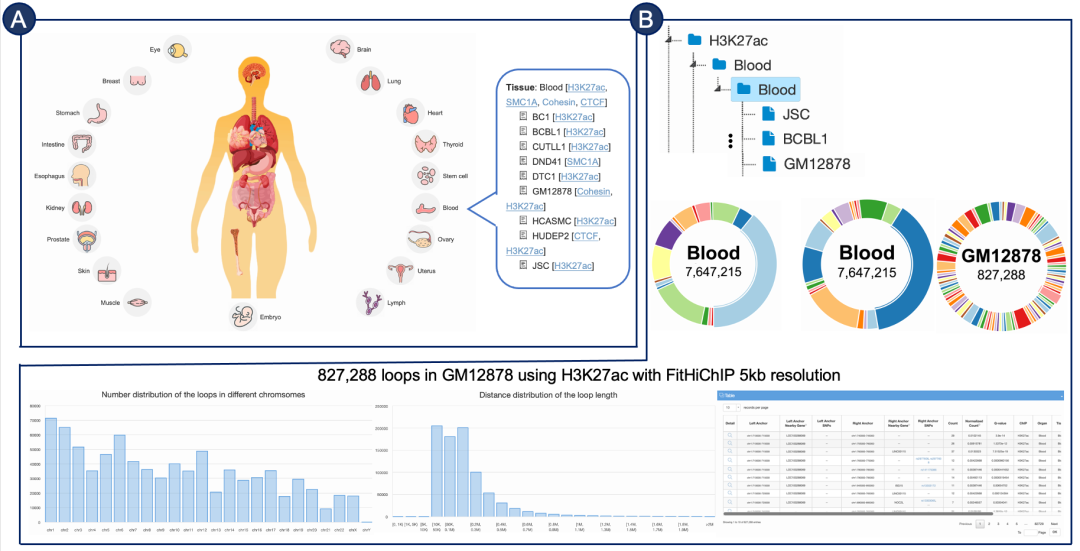
科普网页链接 《测序中国》公众号: https://mp.weixin.qq.com/s/GDpZ7JBK1mAN6hKxtSdP3Q
24. 药物靶标间互作预测的通用图网络框架DeepDrug(江瑞团队)
2023年1月3号,清华大学自动化系江瑞教授团队与斯坦福大学统计系曾婉雯和刘桥博士共同在Quantitative Biology期刊上发表了最新研究成果。他们开发了一个深度学习框架DeepDrug,通过使用残差图卷积网络(Res-GCNs)和卷积网络(CNNs)来学习药物和蛋白质的融合结构和序列信息的全面表示,并在一系列任务(DDI和DTI)中取得最优性能。
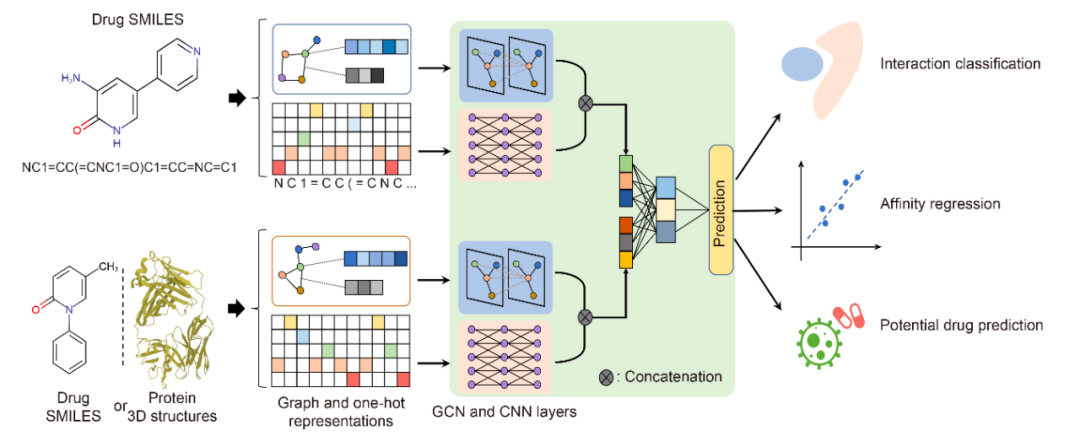
科普网页链接 《QB期刊》公众号: https://mp.weixin.qq.com/s/SRYJVyYHaDMs4CeEBMyLoQ
25. 变分图卷积自编码器框架PAST(江瑞团队)
2023年10月30日,清华大学自动化系江瑞课题组和南开大学数学科学学院陈盛泉课题组在Genome Research上发表了最新研究成果。研究提出了融合生物先验信息和空间位置信息来解析空间转录组(Spatial Transcriptomics)数据、提取低维表示特征的变分图卷积自编码器框架PAST。该模型不仅可以有效提取空间转录组数据的低维表示特征,辨识空间功能区域(Spatial Domains),还能够促进数据可视化、发育轨迹推断和伪时序分析等多种下游任务。此外,PAST还能够进行多切片共嵌入降维分析,进而完成数据整合与标签迁移,促进空间区域自动注释。
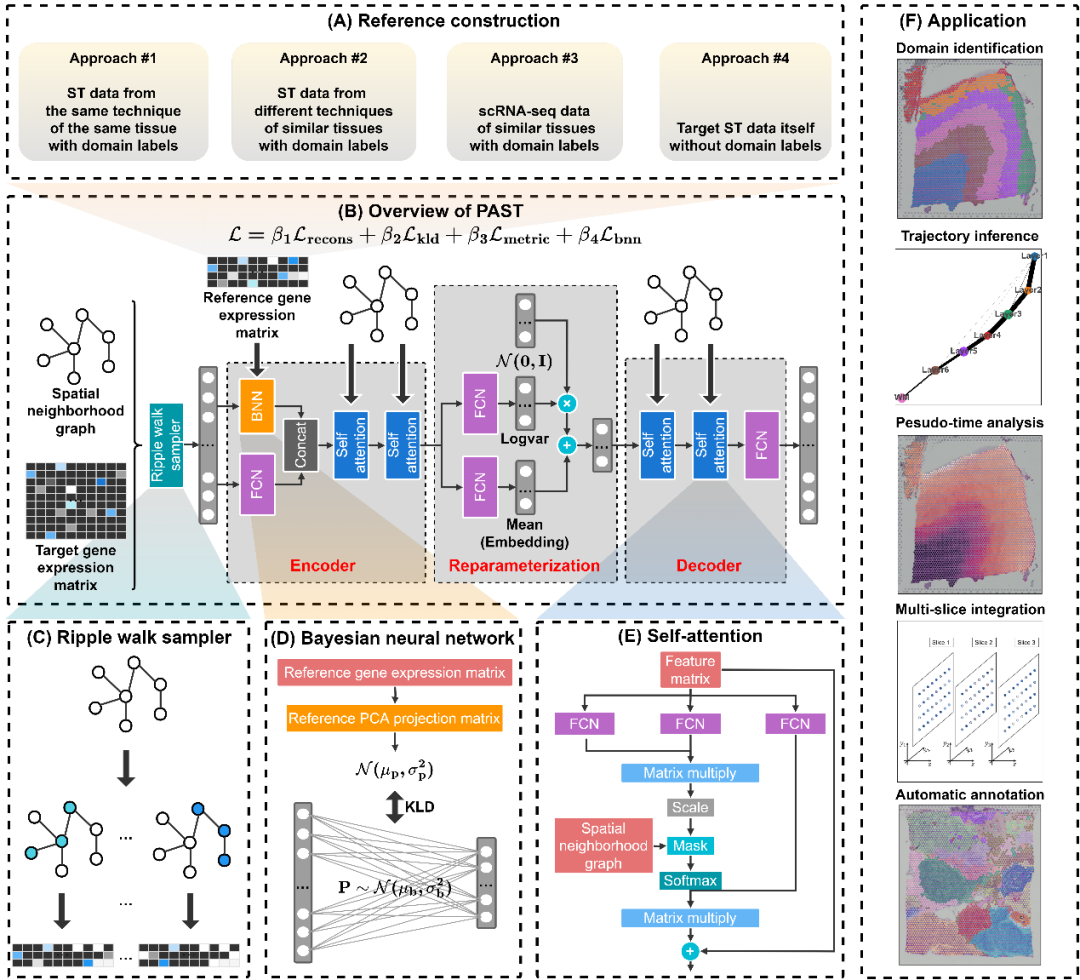
科普网页链接 《测序中国》公众号: https://mp.weixin.qq.com/s/7A6mnnhvRDFLywIPaGENrw
26. 全新单细胞数据分析方法scDEC(江瑞团队)
2021年5月10日,清华大学自动化系的江瑞团队和斯坦福大学统计系的Wing Hung Wong团队合作在国际著名期刊Nature Machine Intelligence发表了最新研究成果。他们提出了全新的单细胞数据分析方法scDEC。 scDEC巧妙地使用一组生成对抗网络将高维单细胞数据映射到低维隐空间,在低维空间进行聚类分析,再使用另一组生成对抗网络将低维数据映射回高维空间。通过这样两组生成对抗网络的往复循环和共同优化,scDEC集细胞类型的辨识与单细胞数据的降维、生成、批次效应去除于一体,不仅实现了融合单细胞基因表达与染色体开放性数据的细胞类型辨识,还成功应用于下游的细胞发育轨迹推断、细胞内基因调控机制解析等细胞功能建模研究。

科普网页链接 《清华大学自动化系》公众号: https://mp.weixin.qq.com/s/LLIdg_P7AgzAg_3EI5UL0w
27. 整合单细胞及群体细胞多组学数据的统一数学框架(江瑞团队)
2019年10月10日,斯坦福大学统计系王永雄Wing H. Wong研究团队与清华大学自动化系江瑞研究团队在Nature Communications上发表最新研究成果。他们将基因调控网络建模推进到单细胞层面,提出了整合单细胞多组学数据和细胞群体组学数据的统一数学框架DC3,克服单细胞层面样本难以匹配的困难,能够对单细胞多组学数据进行更准确的聚类。同时突破三维基因组学数据难以在单细胞层次观测的瓶颈,实现对群体数据进行解卷积分解,计算结果得到实验验证。

科普网页链接 《BioArt》公众号: https://mp.weixin.qq.com/s/MxKc6xjvlV2TcEaPk0jGeQ
28. 概率密度估计的深度生成神经网络方法(江瑞团队)
2021年4月8日,斯坦福大学统计系王永雄Wing H. Wong研究团队与清华大学自动化系江瑞研究团队在《美国科学院院刊》(PNAS)发表最新研究成果。他们提出了基于循环生成对抗模型的Roundtrip方法用于通用性的概率密度估计问题。与以往研究着重于对基本密度和目标密度之间的可逆转换建模不同,Roundtrip允许直接使用深度生成网络来对从隐空间到原始数据空间的转换进行建模,这种思路使得对模型结构与低维隐空间的限制更小、使用更为灵活。
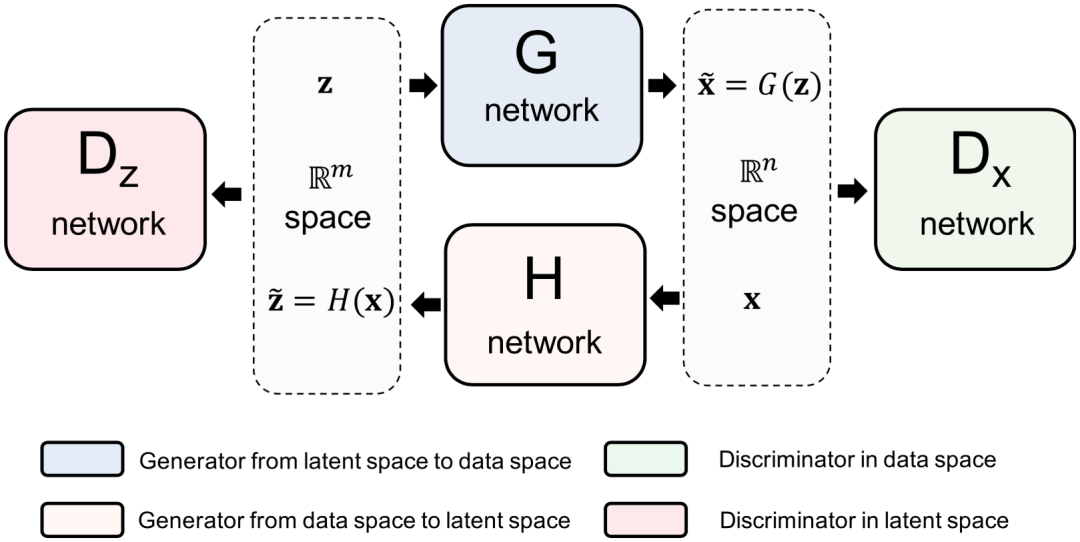
科普网页链接 《集智俱乐部》公众号: https://mp.weixin.qq.com/s/xopC36nkOZfj16Q0gp9xbg
29. 基于参考数据的单细胞表观基因组数据分析模型(江瑞团队)
2021年4月12日,清华大学自动化系的江瑞团队和香港中文大学统计系的林志翔团队合作在Nature Communications发表了最新研究成果。他们利用已有的细胞群测序数据或scCAS数据作为参考数据,研究团队提出了RA3这一全新的单细胞表观基因组数据统计建模方法,从而能够有效地刻画细胞异质性并成功应用于细胞发育轨迹推断和基序富集等下游分析。
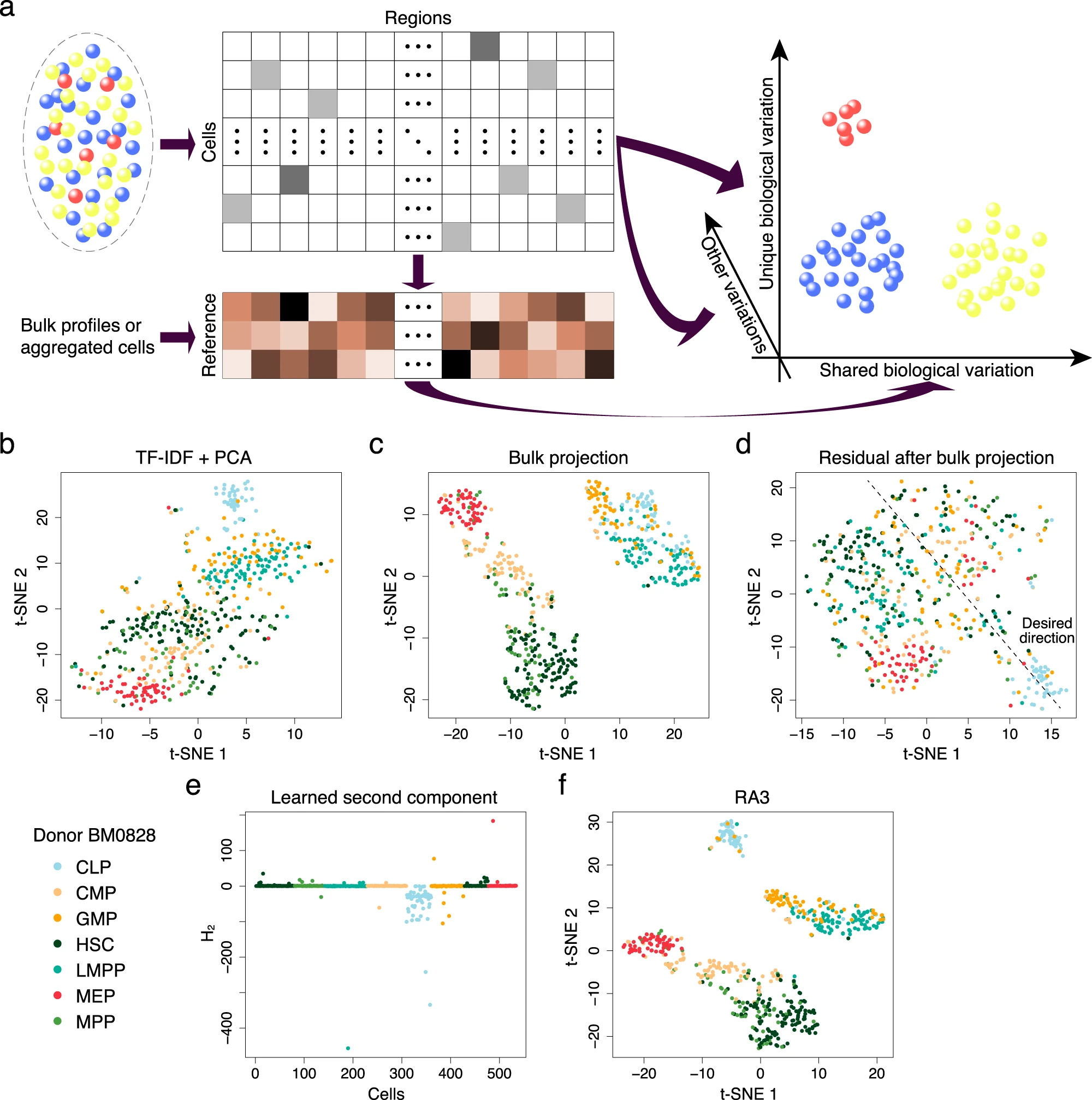
科普网页链接 《BioArt》公众号: https://mp.weixin.qq.com/s/C55MpcX6vkYzjFp4aiI84g
